RNA测序(RNA-seq)是使用深度测序技术来研究生物体转录组的方法,主要包括转录组测序,小RNA测序及基因表达谱测序。数据的单个碱基的分辨率提供了关于单核苷酸多态性(SNP)、选择性剪接、外显子/内含子边界、非翻译区及其它元件的详细信息。RNA-seq不需要预先知道参考序列,使得从头的转录组分析和新的变异体和突变体的检测成为可能。
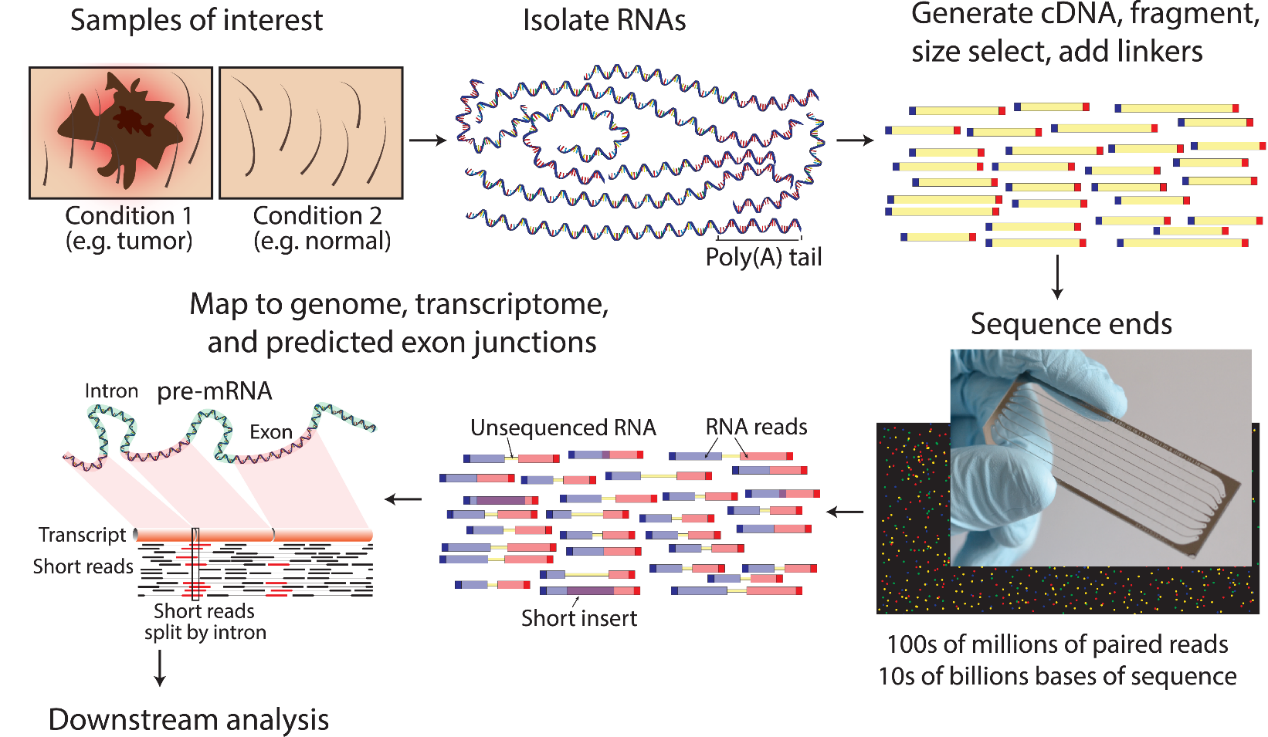 |
| 图 RNA-seq 实验流程 |
RNA-seq中需要考虑的因素
第一个需要考虑的因素是样本富集。总RNA通常只包含比例很少的编码RNA或者功能RNA;样本中大部分RNA是核糖体RNA(rRNA:大约占到了总RNA的80–90%)和较少的转运RNA (tRNA)。为了避免将80–90%的测序资源用在重复的rRNA序列上,通常在测序之前,需要从样本中去除rRNA。这通常可以通过特定的消耗rRNA实现(同时保留编码RNA和非编码RNA),也可以通过使用寡聚胸腺嘧啶富集技术选择性富集Poly A实现(仅保留编码mRNA)。有一些其他的方法可以避免rRNA的影响,比如选择性降解大量转录物,或者不扩增rRNA的扩增技术。但是这些方法不像rRNA消耗或者poly A富集那么常见,并且可能扭曲转录物表征的正常水平。
另外一个需要考虑的因素是要研究的RNA的大小。RNA转录物跨越比较大的大小范围;与常规的RNA分析相比,关注小RNA的实验(比如microRNA或者长度范围在15–35bp的RNA),需要特别的纯化和文库构建方案。大多数其他大小的RN**段可以同时测序(RNA测序的常用步骤之一是将RNA分割成普通长度,比如200–300 nt长度的片段)。
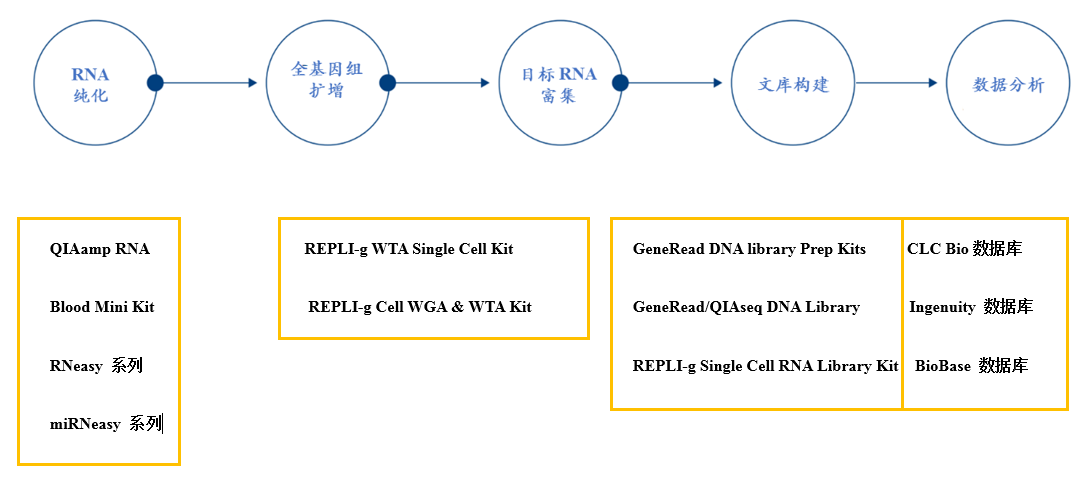
QIAGEN RNA-seq整体解决方案