胚胎干细胞作为一种全能性细胞,通过增殖和分化,产生动物体所有组织和器官的细胞。已有研究表明,胚胎干细胞发生m6A RNA甲基化,大多与细胞增殖[1-2],免疫应答[4]关系密切。然而,对于m6A修饰在胚胎干细胞向神经内胚层细胞分化过程中的分子机制目前并没有相关报道。今天,分享一篇英国剑桥大学研究团队于2018年8月发表在Nature(影响因子:40.14)文章,聊一聊RNA甲基化在调控胚胎干细胞分化过程中的作用机制。这篇文章的开头很直接,首先通过阅读文献确定了感兴趣的基因SMAD2/3,对其进行Co-IP和质谱后,结果显示其能够与甲基化转移酶METTL3,METTL14和WTAP直接互作。他们是明星,和他们结合的分子也是明星分子,这不,在胚胎干细胞当中有研究者就发现SMAD2/3就和METTL3,METTL14结合,不出所料,这一结合在胚胎干细胞当中起到了重要作用。

1.SMAD2/3蛋白与METTL3-METTL14-WTAP复合物结合
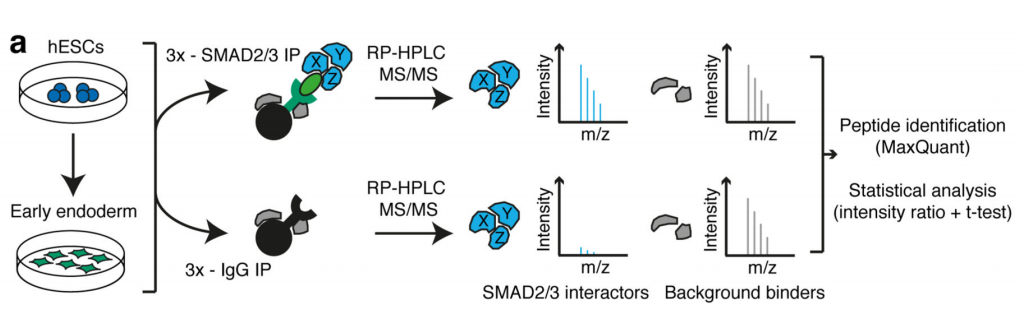
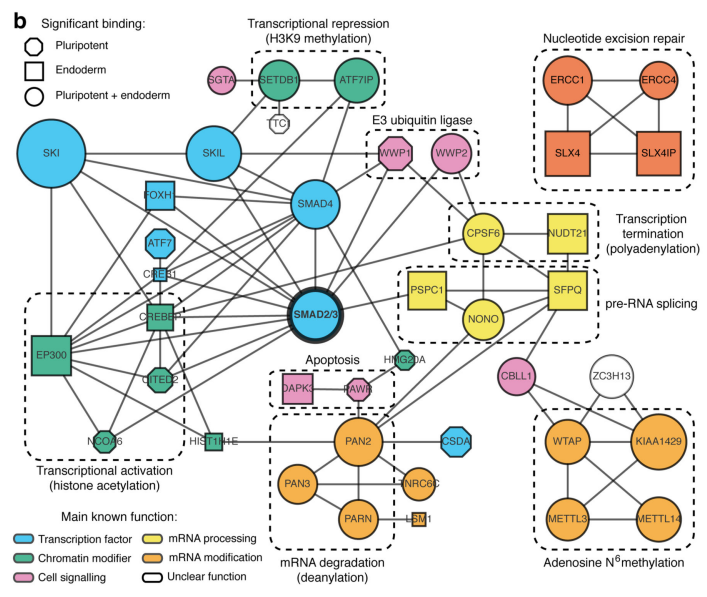
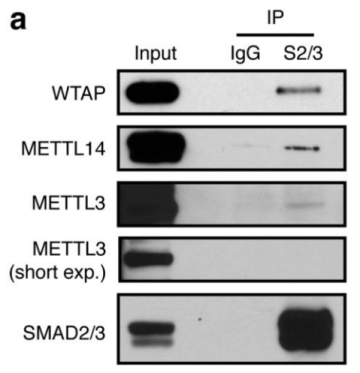
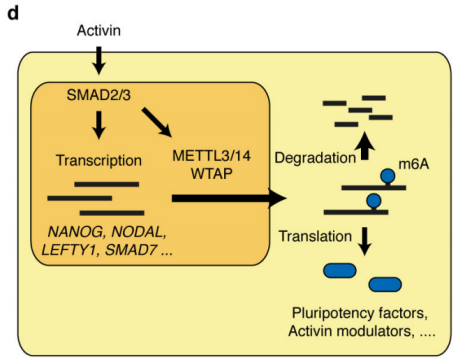
作者首先通过体外刺激,诱导人类胚胎干细胞分化为神经外胚层细胞,并通过Co-IP技术联合质谱(云序生物提供此项实验)技术,对比两种细胞中,能够与SMAD2/3抗体直接结合的蛋白。结果显示,一共找到89个蛋白,取交集后,得到78个共有蛋白,绘制成网络图,发现这些蛋白除参与TGF-β信号通路,mRNA生物学过程之外,还与国自然大热点RNA甲基化中的甲基化转移酶-------METTL3-METTL14-WTAP复合物直接互作。此结果与后期WB验证和抑制SMAD2/3磷酸化位点后Co-IP结果吻合。PLA实验证实两者间的结合主要发生在细胞核内,并且受Activin刺激后,影响核内转录因子NANOG表达。
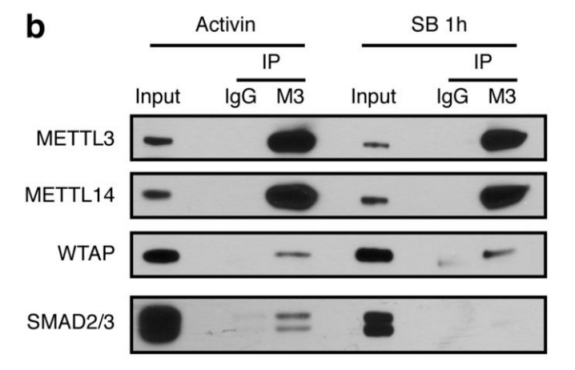
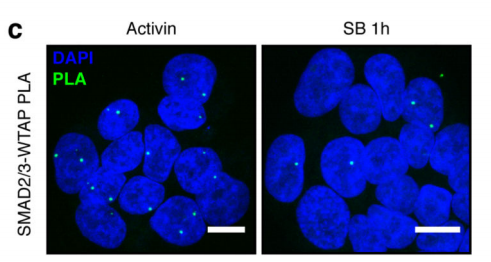
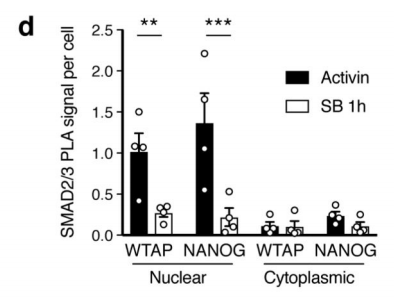
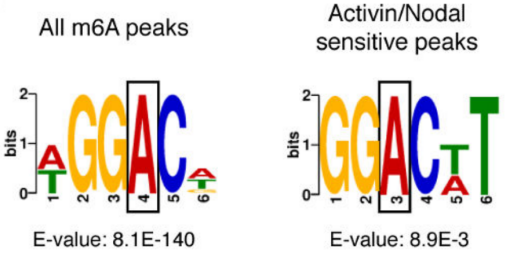
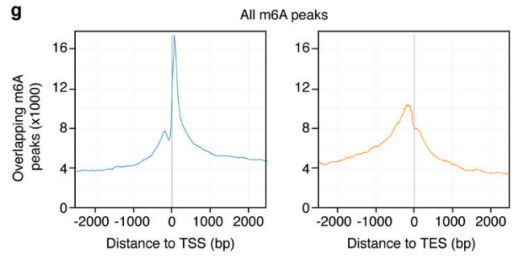
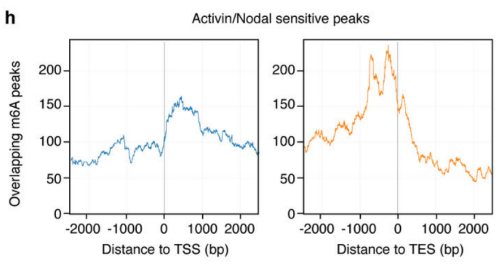
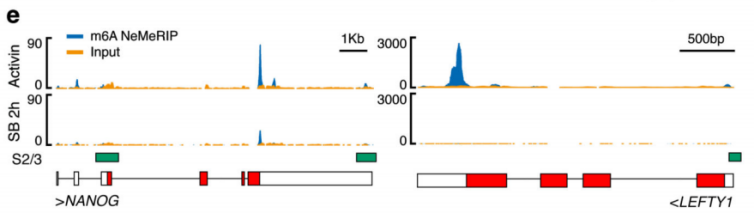
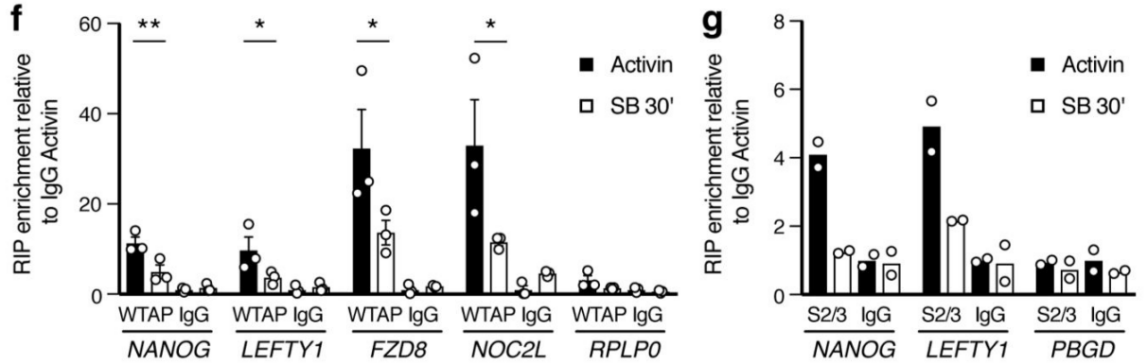
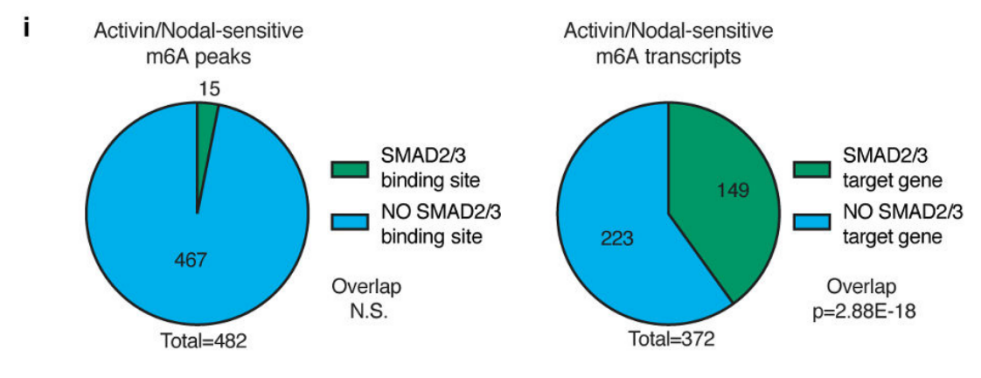
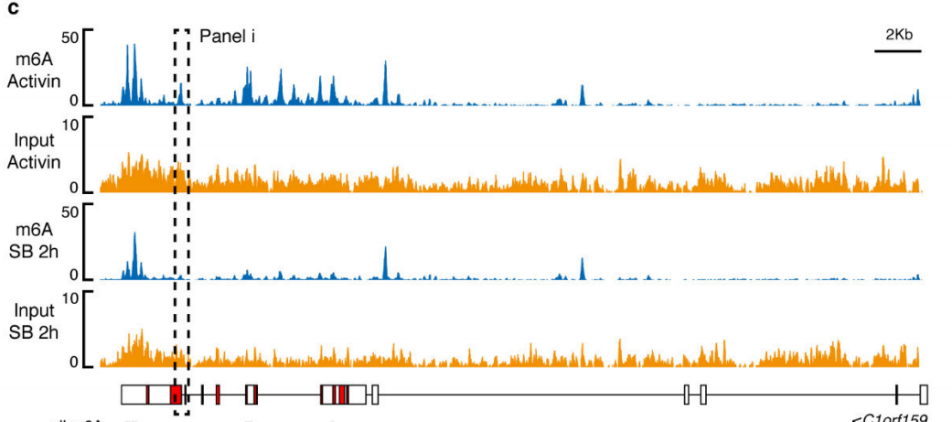
作者以Activin/Nodal敲降组胚胎干细胞为实验组,未敲降组为对照,每组三个生物学重复,进行m6A RNA甲基化测序(云序生物提供此项实验)。结果显示,motif区域保守序列为GGAC,且大多在转录起始区和转录终止区富集,Activin/Nodal特有甲基化位点在转录起始区低富集。可视化结果显示,NANOG和LEFTY1的mRNA分子上存在m6A甲基化信号(如红框所示),并且通过SMAD2/3的ChIP测序(云序生物提供此项实验)结果,有一部分区域与甲基化信号重叠(如绿框所示)。WTAP或SMAD2/3蛋白RIP实验(云序生物提供此项实验)证实,在受Actin分子刺激下,WTAP与NANOG,LEFT,FZD8和NOC2L结合增强,而在SMAD2/3分子刺激下,能够促进其降解。
前人研究证实m6A多在细胞核内促进pre-RNA的合成,由于本实验的SMAD2/3通过与m6A 甲基化酶复合物结合,影响靶基因合成的位置同样也在核内,并且SMAD2/3的转录和甲基化受Activin/Nodal调控,因此,作者猜测SMAD2/3可能参与pre-mRNA的合成。通过抑制Activin/Nodal后检测m6A RNA甲基化水平,发现差异甲基化的情况不单单在外显子上下调,同时在外显子和内含子交界处受抑制。综上,Actin通过促进磷酸化的SMAD2/3蛋白与下游转录因子结合,调控靶基因发生m6A RNA甲基化。
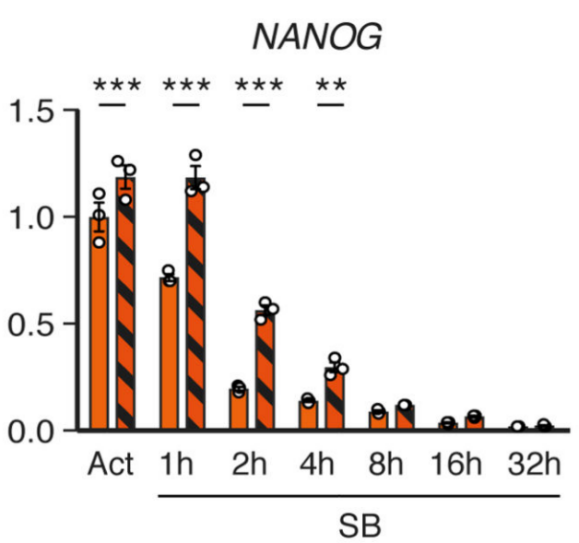
通常来说,RNA甲基化与基因的表达关系密切。作者接下来分析了甲基化与转录水平的两组学联合分析,发现下调Activin/Nodal通路后,NNANOG上的m6A甲基化水平与表达量成反比,并对神经内胚层分化起促进作用。
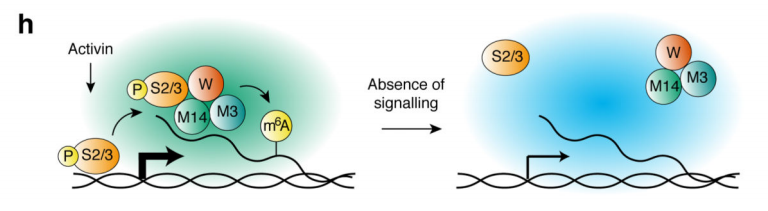
本篇文章首次研究了SMAD2/3蛋白与TGF-β通路间复杂的细胞内生物学过程。本文结果展示了SMAD2/3在mRNA上发挥了调控,凋亡,DNA修复和转录后调控的生物学功能,并阐述了此过程受Activin/Nodal的m6A甲基化调控,并与甲基化转移酶间的关联。本文为以后研究RNA甲基化非经典机制,如与染色质表观遗传、转录和表位调控奠定基础。
全文链接
https://www.nature.com/articles/nature25784
1.Yang D, Qiao J, Wang G, et al. N6-Methyladenosine modification of lincRNA 1281 is critically required for mESC differentiation potential[J]. Nucleic Acids Research, 2018, 46. 影响因子:11.561
2.Wen J, Lv R, Ma H, et al. Zc3h13 Regulates Nuclear RNA m6A Methylation and Mouse Embryonic Stem Cell Self-Renewal.[J]. Molecular Cell, 2018, 69(6):1028. ?影响因子: 14.248
3.Li H B, Tong J, Zhu S, et al. m6A mRNA methylation controls T cell homeostasis by targeting the IL-7/STAT5/SOCS pathways[J]. Nature, 2017, 548(7667):338-342. 影响因子: 41.577?
云序生物RNA甲基化产品列表
云序相关产品推荐
往期回顾
Nat. commun|Mettl3介导的m6A RNA甲基化调控骨髓间充质干细胞和骨质疏松症
Nature|m6A RNA甲基化识别蛋白YTHDF1参与记忆的形成
Plant Cell:拟南芥发现全新m6A RNA去甲基化修饰酶
云序客户12分顶级文章,教你如何巧用RIP测序玩转分子机制!
2018年Nature杂志重磅级突破性研究成果--m5C RNA甲基化 云序生物
Nature重磅:m6A RNA甲基化参与造血干细胞发育关键环节!
Cancer Cell: 何川教授在发现m6A RNA甲基化重要功能
Hepatology:m6A RNA甲基化酶METTL3促肝癌发展新玄机
地 址: 上海市松江区莘砖公路518号24号楼4楼 联系人: 戴小姐 电 话: 021-64878766 传 真: 021-64878766 Email:market@cloud-seq.com.cn;liuqingqing@cloud-seq.com.cn
上海云序生物科技有限公司
商家主页
相关咨询
杨宝峰院士团队RNA修饰又一成果 | 云序ac4C acRIP-seq助力揭示心脏I/R损伤的作用机制 (2024-12-03T00:00 浏览数:12725)
杨宝峰院士团队最新成果 | 云序助力揭示RNA修饰m7G调控心肌肥厚的机制研究 (2024-11-13T00:00 浏览数:14513)
Nature子刊| 重磅综述!一文总结「m6A修饰非编码RNAs」在各类肿瘤中的调控机制及作用 (暂无发布时间 浏览数:11494)
研究速览-eccDNA 2023年最新进展大放送! (暂无发布时间 浏览数:12731)
云序生物MeRIP-qPCR技术干货 (暂无发布时间 浏览数:12643)
技术干货| “eccDNA碱基序列的获取及引物设计”方法教程 (暂无发布时间 浏览数:11815)
云序客户m6A高分文章|揭示组蛋白乙酰化与m6A修饰在眼部黑色素瘤发生中的共同作用机制 (暂无发布时间 浏览数:6501)
Nat Biotechnol IF=47 | BID-seq:一种基于单碱基分辨率的假尿嘧啶(Ψ)修饰定量测序检测方法 (暂无发布时间 浏览数:6417)
北大伊成器团队Nature Reviews重磅发文:非m6A热门修饰调控与功能一文速览! (暂无发布时间 浏览数:10076)
用户文章m6A专题|IF=9.8|m6A去甲基化酶ALKBH5缺乏会加重钴致神经退行性损伤 (暂无发布时间 浏览数:6380)
