数据归一化
多维统计模型建立之前,首先需要对数据作归一化(Normalization)处理(有些学者称为标准化Standardization),一是让数据无量纲化,使不同性质的变量具有可比性;二是将不同数量级的变量数据经过不同的转换(transform)至合适范围,避免大值变量掩盖小值变量的波动。在代谢组学数据处理中,常用的归一化方法有Ctr(Center scaling), UV (unit variance scaling)和Par(Pareto scaling)。Ctr也叫中心化是原数据减去每列变量的均值,UV是数据中心化后除以列变量标准差(Standard deviation),Par是数据中心化后除以列变量标准差的算术平方根。Ctr将原数据转化成离原点更近的新数据,可调节代谢物的高低浓度差异;UV的优势是所有变量拥有同等的重要性,但缺点是检测误差可能会被放大;Par相比于UV更接近于原始测量数据,但缺点是对变化倍数大的变量更敏感[1]。UV和Par是常用的归一化方式,基于不同的归一化方式后续的数据分析将选择不同的差异代谢物筛选方法,如UV下常使用V-plot(图1-A),Par下则常用S-plot(图1-B)。无论选择何种归一化方式,都需要对建立的模型作严格验证以确保筛选出可靠的差异代谢物。因为VIP值通常用于差异变量筛选标准之一,V-plot可比较客观的选择出变量。对于Biomarker Discovery的诊断工具,我们推荐使用V-plot和相关性Corr.Coeffs. 的p值同时考虑的标准,如下图2所示。
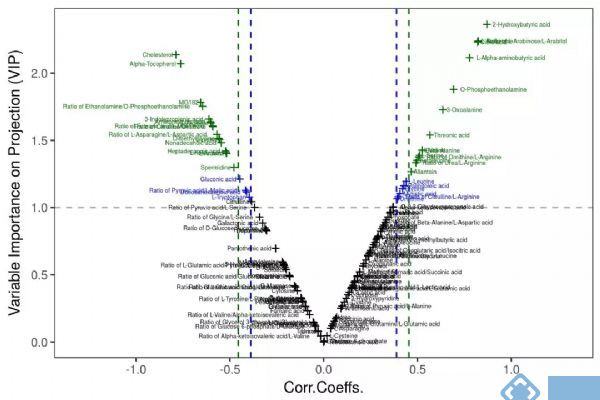
图1. V-plot和S-plot示意图
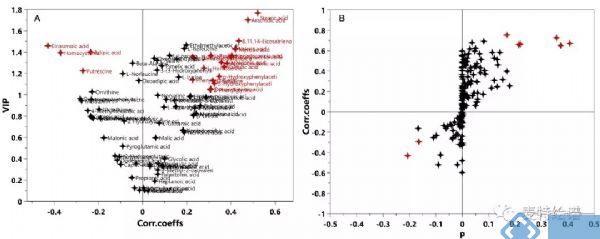
图2. 基于VIP和Corr.Coeffs的p值的V-plot用于差异代谢物的筛选。来源:麦特绘谱XploreMET软件。
模型构建
代谢组学数据分析中,最常用的多维模型包括主成分分析(principal component analysis, PCA)、偏最小二乘法判别分析(Partial least squares discriminant analysis, PLS-DA)和正交偏最小二乘法判别分析(orthogonal PLS-DA, OPLS-DA)。PCA属于无监督的分类模型,可将多维的数据不断降维形成几个主要成分(PC)来尽可能描述原始数据的特征。其中PC1描述了原始数据矩阵中最显著的特征,PC2描述了除PC1之外最显著的数据特征,依此类推。PCA通常被用于寻找离群点(outlier)及观察不同组别之间的自然聚类趋势。那么如何判断数据集中的outlier?可通过Hotelling's T2或PC1的score plot(PC1的数据解释率最高)来判断(图3),通常红线之外的样本为严重离群点,需要进一步处理。PCA的离群点也可以分组来看,以减少组间的干扰,如下图4所示。但对于离群点,不建议简单粗暴地删除,因为离群点通常是有趣且值得深究的。研究人员需要仔细地排查离群究竟是因为采样、前处理、检测等环节引入的误差还是客观的生物学差异引起的。
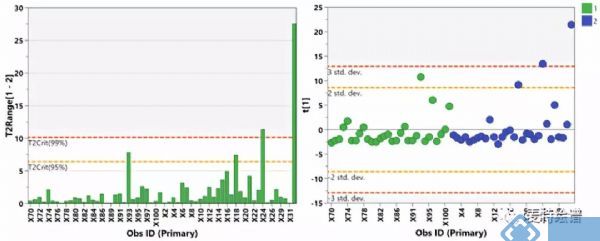
图3. Hotelling's T2柱状图和PC1的得分图
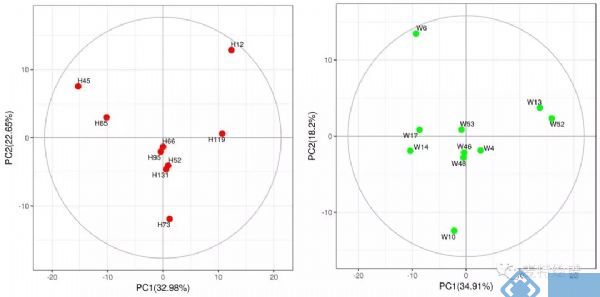
图4. 分组PCA 得分图用于离群点寻找。来源:麦特绘谱XploreMET软件。
利用PCA模型还可以观察样本间的自然聚类趋势。不同组别样本在PCA Score plot上即可分离是多维统计结果可靠性的最有力证据。然而,不同组别样本不一定都存在明显的差异,尤其对于临床样本的影响因素较多,如性别、年龄、BMI、地域、饮食、生活环境等。这些因素会给数据集带来很多和分组信息无关的噪音信号。此时,可以利用有监督的分类模型。有监督的意思就是事先告诉模型样本的真实分组信息再进行模型构建。PLS-DA能按照预先定义的分类(Y变量)最大化组间的差异,获得比PCA更好的分离效果(图5)。OPLS-DA综合了PLS-DA和正交信号过滤(orthogonal signal correction, OSC)技术,能够把与预先设定的和分类无关的信息最大程度从原始矩阵分离,从而将最相关的因素集中到第一个主成份(Predictive component)上,进而寻找该主成分的正交矫正轴方向,从而使得组间样本分离效果更佳,使组内差异弱化,组间差异最大化凸显,且更适用于两组样本间的分离。PLS-DA可以用于两组及以上组别的分类比较,而OPLS-DA通常用于两组的对比,找差异物质。
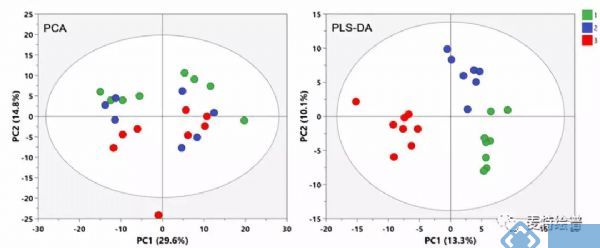
图5. PCA和PLS-DA得分图,PLS-DA可获得更清晰的分离
模型评价
有监督的分类模型缺点是可能会出现过拟合(over-fitting)现象,即模型可以很好地将样本进行区分,但用来预测新的样本集时却表现很差。因此对于有监督的分类模型,我们需要验证模型的可靠性,下面列出几种常见的模型评价方法:
1. K折交互验证(K-fold cross validation)
最可靠的方式是将数据分为训练集(Training set)、验证集(Validation set)和测试集(Test set),训练集用于训练模型,验证集优化模型,测试集测试模型的预测能力。但受限于样本数量,通常采用K折交互验证。其中七折交互验证较为常用,即将数据集分为7份,每次挑选出1份作为测试样本,剩余的6份用来训练建模,整个过程将会被重复直到所有样品都被预测过。预测的数据将会和原始数据作对比得到预测残差平方和(Predicted residual sum of squares, PRESS)。为方便起见,将PRESS转变为Q2(1-PRESS/SS)。Q2越大表示模型的预测能力越好。对于生物学样本,Q2≥0.4是比较理想的[2],Q2≥0.2往往也可以接受,只是模型比较弱。软件在自动建模(Autofit)时,会根据Q2决定模型所用的主成分或Orthogonal component个数(OPLS-DA模型)。当Q2停止增长时,模型将不再增加主成分。
2. 置换检验(Permutation test)
仅用Q2仍不足以证明模型的可靠性,置换检验也是常用的模型评判方式,常和Q2结合使用。其原理是将每个样本的分组标记随机打乱,再来建模和预测。一个可靠模型的Q2应当显著大于将数据随机打乱建模后得到的Q2。基于置换检验的结果,可以画出Permutation plot(图6)。该图展示了置换检验得到的分组变量和原始分组变量的相关性以及对应的Q2值,虚线为回归线。一个可靠的有监督模型要求回归线在Y轴上的截距小于0。
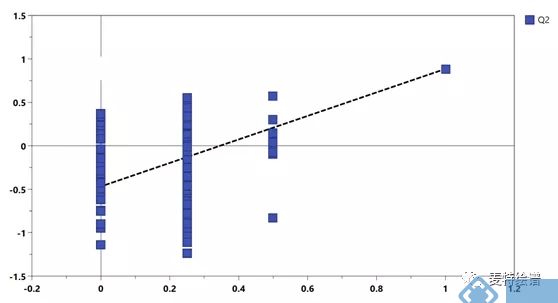
图6. Permutation plot用于模型验证
3. 基于交互验证的方差分析(CV-ANOVA)
CV-ANOVA是基于交互验证预测残差的方差分析,利用方差分析测试预测的Y变量(Yhat)和预设Y变量(Yobs)的残差和Yobs围绕均值变化的差异。它的好处是可以将交互验证的结果以更加熟悉的方式展现出来,输出表征统计学意义的P值。但CV-ANOVA对于小样本集的检验效能较低[3]。
差异代谢物筛选
筛选差异代谢产物通常基于OPLS-DA模型,因为它更易于进行模型解释,所有跟分组相关的信息都集中于第一维。筛选的标准通常是基于以下两个指标:
Corr.Coeffs./p(corr) (Correlation Coefficient),是样本得分值t和变量X间的相关系数-Corr(t, X),代表了变量的可靠度。该值没有固定阈值,通常设定对应的P值 < 0.05。
VIP (Variable importance in the projection),为变量对模型的重要性,描述了每一个变量对模型的总体贡献,通常设定阈值为VIP >1。
除此之外,基于单维检验的P值和变化倍数(Fold change)所作的火山图(Volcano plot)也是常用的筛选方法。
代谢通路分析
通过上述方法筛选到差异代谢物后,还需要挖掘和这些代谢物相关的代谢通路。此时,可以采用MetaboAnalyst网站(http://www.metaboanalyst.ca/)进行代谢通路分析(Metabolic pathway analysis),代谢通路分析分为富集分析(Enrichment analysis)和通路分析(pathway analysis)。通路分析中添加了通路拓扑分析(topology analysis),会输出通路在整体网络中的重要性(impact)。下图展示了典型的代谢通路分析图。
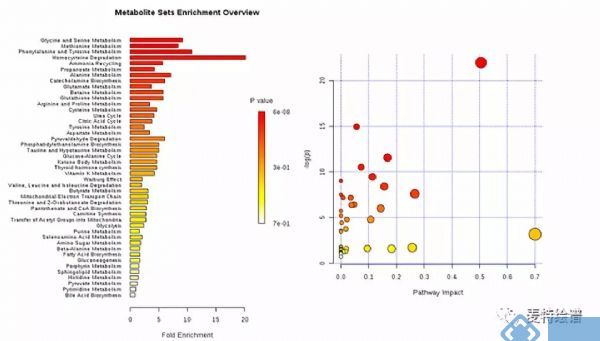
图7. 代谢通路分析展示图(来源:MetaboAnalyst网站)
代谢组学的数据处理远不止于此,本文浅尝辄止,若有更多感兴趣的问题,请在下方留言,欢迎一起交流讨论!
参考文献
Worley, B. and R. Powers, Multivariate Analysis in Metabolomics. Curr Metabolomics, 2013. 1(1):92-107.
Westerhuis, J.A., et al., Assessment of PLSDA cross validation. Metabolomics, 2008. 4(1):81-89.
Eriksson, L., J. Trygg, and S. Wold, CV-ANOVA for significance testing of PLS and OPLS® models. J. Chemometrics, 2008. 22:594–600.
